Antecedentes: La miocardiopatía hipertrófica (MCH) es una enfermedad genética del corazón y la causa más común de muerte cardíaca súbita en los jóvenes. La MCH se considera una enfermedad del sarcómero debido al gran número de mutaciones en los genes que codifican las proteínas sarcoméricas. El enigma reside en descubrir cómo estas mutaciones conducen a la enfermedad. Por ello, los tratamientos para prevenir y/o tratar la MCH se limitan a miectomías o ablaciones quirúrgicas invasivas. La variante A31P de la proteína C de unión a la miosina cardíaca, codificada por MYBPC3, resultó ser más prevalente en una cohorte de gatos Maine Coon con MCH. Sin embargo, otras mutaciones en MYBPC3 y MYH7 también se han asociado a la MCH en gatos de otras razas. En este estudio, ampliamos el espectro de genes asociados a la MCH en gatos.
Resultados: Se llevó a cabo la secuenciación del genoma completo de próxima generación utilizando el ADN aislado de la sangre periférica de un Maine Coon con cardiomiopatía que resultó negativo para la variante MYBPC3 A31P. Mediante la estratificación del riesgo de las variantes, identificamos una nueva variante intrónica homocigótica en la troponina T cardíaca (TNNT2). El análisis in silico de la variante sugirió que podría afectar al empalme normal del exón 3 de la TNNT2. Ambos padres resultaron heterocigotos para la mutación, pero no estaban afectados por la enfermedad. Los análisis ecocardiográficos revelaron que el probando había mostrado una insuficiencia cardíaca congestiva de aparición temprana, que se controla con un régimen de tratamiento que incluye inhibidores de la ECA y de la aldosteronaConclusión: En resumen, somos los primeros en demostrar la asociación entre las mutaciones de TNNT2 y la MCH en felinos, lo que sugiere que este gen debería incluirse en el panel de pruebas de genes cuando se realicen pruebas genéticas para la MCH en gatos.
Conclusión: En resumen, somos los primeros en demostrar la asociación entre las mutaciones de TNNT2 y la MCH en felinos, lo que sugiere que este gen debería incluirse en el panel de pruebas de genes cuando se realicen pruebas genéticas para la MCH en gatos.
Introducción
La miocardiopatía hipertrófica (MCH) es la forma más común de cardiopatía genética y afecta al menos a 1 de cada 500 seres humanos (Ho, 2011). Definida por un inexplicable engrosamiento anormal de las paredes ventriculares y una posible obstrucción del tracto de salida, actualmente no existe cura para la MCH. La MCH está causada principalmente por mutaciones en los genes que codifican las proteínas sarcoméricas. En particular, las mutaciones en MYH7 y MYBPC3, que codifican la cadena pesada de la miosina cardíaca y la proteína de unión a la miosina cardíaca-C, respectivamente, son los genes mutados con mayor frecuencia (Viswanathan et al., 2017), aunque otros genes sarcoméricos, incluidos TNNI3, TNNT2 y MYL2, también se han implicado en humanos (Brouwer et al., 2011). Todavía no se sabe cómo las mutaciones en estos genes sarcoméricos resultan en el desarrollo de la MCH.
En los gatos, la cardiomiopatía es la principal causa de enfermedad cardiovascular, siendo la MCH la forma más común, lo que sugiere que los gatos pueden ser un excelente modelo animal no roedor de MCH (Ferasin et al., 2003). Una variante en MYBPC3, A31P, se relacionó previamente con el desarrollo de MCH en gatos Maine Coon (Meurs et al., 2005). Esta variante se produce dentro del dominio C0 de la proteína C de unión a la miosina cardíaca (cMyBP-C), y se espera que cause la enfermedad a través de defectos estructurales en este dominio importante para la regulación de la actomiosina (Van Dijk et al., 2016). Otra variante en MYBPC3, A74T, también se ha descrito en múltiples razas de gatos. Sin embargo, estudios de seguimiento en profundidad demostraron que este polimorfismo no está relacionado con la cardiomiopatía (Wess et al., 2010; Longeri et al., 2013). Una tercera variante en MYBPC3, R820W, se ha asociado con la MCH, más específicamente en el Ragdoll (Meurs et al., 2007). Más recientemente, la primera variante en MYH7, E1883K, se ha asociado con la MCH en un gato doméstico de pelo corto, ampliando el espectro de genes asociados con la cardiomiopatía felina (Schipper et al., 2019). En la Tabla 1 se muestra una lista completa de mutaciones asociadas a la cardiomiopatía felina.

Tabla 1. Lista actual de mutaciones conocidas asociadas a la cardiomiopatía felina.
En el presente estudio, se estudió un gato Maine Coon macho con cardiomiopatía e insuficiencia cardíaca congestiva temprana. Tras un cribado inicial negativo para la mencionada mutación A31P, realizamos la WGS para identificar posibles mutaciones y, así, precisar la causa genética de la cardiomiopatía de este gato mediante el estudio del árbol genealógico del gato enfermo. Este estudio amplía la reciente secuenciación de próxima generación realizada en gatos (Genova et al., 2018; Ontiveros et al., 2018). Como resultado, hemos identificado una nueva mutación autosómica recesiva en el gen de la troponina cardíaca sarcomérica T (TNNT2) que parecía estar asociada a la cardiomiopatía felina. Este es el primer registro conocido de tal mutación en felinos, lo que sugiere que TNNT2 debe incluirse como un gen candidato en los cribados genéticos.
Materiales y Métodos
Fenotipado, recogida de sangre y extracción de ADN
Todos los procedimientos siguieron el protocolo aprobado por el Comité Institucional de Cuidado y Uso de Animales de la Universidad de Cincinnati y cumplieron con la Guía para el Uso y Cuidado de Animales de Laboratorio publicada por los Institutos Nacionales de Salud. El propietario del probando estableció contacto para determinar si la enfermedad del animal podía estar relacionada con una mutación genética. Para establecer un estudio de investigación completo, se inició un acuerdo de transferencia de material entre el propietario del probando y la Universidad de Cincinnati. Los investigadores no tuvieron contacto con ningún animal más allá de la recepción de muestras de sangre. Todos los estudios que implicaban contacto con los animales fueron realizados por veterinarios certificados. El diagnóstico y el tratamiento del probando, las revisiones periódicas y la ecocardiografía fueron realizados por un cardiólogo veterinario certificado utilizando técnicas ecocardiográficas estándar y referencias de una población de gatos Maine Coon sanos (Thomas et al., 1993; Drourr et al., 2005). La sangre periférica fue recogida por un veterinario certificado o una enfermera veterinaria en el hospital local de cada animal tras una revisión rutinaria. La sangre recogida localmente se transportó al laboratorio en hielo húmedo. La sangre que se recogía en clínicas veterinarias más alejadas del laboratorio se enviaba en bloques fríos durante la noche. Una vez recibidas en el laboratorio, todas las muestras de sangre se alicuotaron en muestras de 200 μl y se almacenaron a -80°C hasta su uso. El ADN se extrajo de una sola alícuota de 200 μl utilizando el kit de aislamiento de ADN QIAamp DNA Mini y Blood DNA de Qiagen, siguiendo el protocolo del fabricante. La concentración y la pureza del ADN se analizaron mediante NanoDrop. En el caso de las muestras enviadas para WGS, la integridad y la concentración del ADN también se determinaron utilizando el bioanalizador Agilent 2100 y el fluorómetro Qubit, respectivamente.
Secuenciación y análisis del genoma completo
El ADN extraído de la sangre total del probando se envió a Novogene Co., Ltd., para la WGS. La calidad y la cantidad del ADN se confirmaron mediante electroforesis en gel de agarosa y fluorómetro Qubit. Una cantidad total de 1,5 μg de ADN genómico de alta calidad se cortó al azar en fragmentos cortos de aproximadamente 350 pb y se utilizó para la construcción de bibliotecas utilizando el NEBNext® DNA Library Prep Kit. Brevemente, tras la reparación de los extremos, la cola dA y la ligadura con el adaptador NEBNext, los fragmentos se enriquecieron por PCR con oligos P5 y P7 indexados. La concentración de la biblioteca de ADN se cuantificó con el fluorímetro Qubit 2.0 y se diluyó a 1 ng/μl. Tras la dilución, se evaluó el tamaño de inserción de la biblioteca con el bioanalizador Agilent 2100 y se realizó una PCR cuantitativa en tiempo real (qPCR) para detectar la concentración efectiva de la biblioteca preparada. Tras el enriquecimiento y la indexación, se realizó la secuenciación por pares de la biblioteca cualificada en un Illumina HiSeq X Ten con una longitud de lectura de PE150bp en cada extremo.
Análisis bioinformático
Los datos de secuenciación originales adquiridos por plataformas de secuenciación de alto rendimiento (por ejemplo, Illumina HiSeqTM/NovaSeqTM) y registrados en archivos de imagen se transformaron primero en lecturas de secuencias mediante la llamada de bases con el software CASAVA. La información sobre las secuencias y la calidad de secuenciación correspondiente se almacenó en un archivo FASTQ. Tras el control de calidad de los datos de secuenciación en bruto para el filtrado de datos limpios, cada lectura limpia se mapeó con el genoma de referencia (Felis_Catus_9.0, Ensembl release 93) utilizando el software BWA (Li y Durbin, 2009), y la tasa de mapeo y la cobertura se contaron de acuerdo con los resultados del alineamiento. Los duplicados se eliminaron con SAMTOOLS (Li et al., 2009). Los polimorfismos de un solo nucleótido (SNP) y las variantes InDel se detectaron con el software GATK (Depristo et al., 2011). Estos SNP se anotaron utilizando ANNOVAR como se describió anteriormente (Wang et al., 2010).
Secuencia de Sanger
Para probar, o confirmar, la presencia de una variante genética en los padres del probando, se realizó una PCR utilizando una polimerasa Taq de alta fidelidad para amplificar específicamente la secuencia alrededor de la variante. El producto de la PCR se purificó y se envió para la secuenciación de Sanger en el centro de secuenciación y genotipado de ADN del Hospital Infantil de Cincinnati.
Resultados
El sujeto de prueba era un macho de raza pura Maine Coon de propiedad privada. A los 8 meses de edad, presentaba una dilatación del ventrículo izquierdo, la aurícula derecha y el borde de la aurícula izquierda, así como una hipertrofia septal límite, con una función sistólica entre preservada y elevada (Figuras 1A,B y Tabla 2), en comparación con los valores de ecocardiografía de referencia para el Maine Coon (Drourr et al., 2005). La función diastólica no pudo cuantificarse como resultado de la fusión de las ondas E y A. A los 14 meses de edad se observó un agrandamiento progresivo de las cuatro cámaras del corazón, mientras que se observó una hipertrofia septal limítrofe y una función sistólica preservada. Se diagnosticó una miocardiopatía primaria no clasificada con posible insuficiencia cardíaca congestiva temprana. El veterinario inició un tratamiento con furosemida, benazepril, espironolactona y clopidogrel. Esta combinación pareció estabilizar la progresión de la cardiopatía a los 18 meses (Tabla 2).
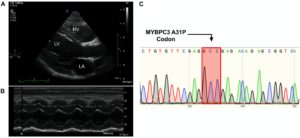
Figura 1. Cardiomiopatía en ausencia de la variante MYBPC3 A31P. (A,B), Ecocardiografía representativa y esquema de estratificación del riesgo de enfermedad cardiovascular de las variantes genéticas. Imágenes representativas en modo B (A) y modo M (B) de eje largo paraesternal del sujeto de prueba, con el ventrículo izquierdo (LV), la aurícula izquierda (LA) y el ventrículo derecho (RV) marcados. (C), El cromatograma de MYBPC3 A31P demostró que el sujeto de prueba era negativo para esta variante.
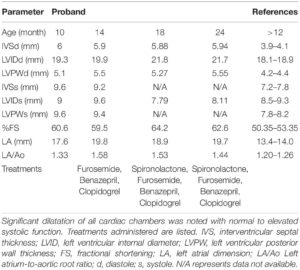
Tabla 2. Parámetros ecocardiográficos de mediciones seriadas realizadas por un cardiólogo veterinario certificado.
Se aisló el ADN de la sangre periférica completa del sujeto. Se realizó una PCR para producir un amplicón de 251 alrededor de la variante A31P en MYBPC3 previamente asociada con la MCH en gatos Maine Coon (secuencias de los cebadores en la Tabla 3). La secuenciación de Sanger del producto de la PCR no mostró ninguna mutación en el codón 31, indicando que el probando no era portador de la variante patógena A31P en MYBPC3 (Figura 1C).

Tabla 3. Lista de secuencias de cebadores utilizadas en este estudio para comprobar la presencia de variantes en los genes MYBPC3 y TNNT2.
Dado que el sujeto era negativo para la mutación A31P en MYBPC3, nos preguntamos si había alguna otra variante que pudiera ser relacionada con la cardiomiopatía del gato. En consecuencia, se aisló el ADN en fresco para la WGS con el fin de identificar posibles variantes presentes en el sujeto. Se envió ADN de alta calidad con un tamaño aproximado de 22944 pb para la preparación de la biblioteca y la secuenciación por Novogene. Las secuencias se alinearon con el genoma de referencia de Felis Catus (Ensemble release 93). La secuenciación dio como resultado 177419470 lecturas en bruto, con una profundidad media de 17,59 lecturas por base, una tasa de error de 0,03 y un 94,19% de lecturas de base con una puntuación Phred superior a 30 (Figuras 2A-C). El contenido de GC detectado fue del 43,06% en comparación con el genoma de referencia, que fue del 41,73%. En conjunto, estos datos demostraron una calidad suficientemente alta de los datos de secuenciación.

Figura 2. Control de calidad de los resultados de la secuenciación de próxima generación. (A) De 177.419.470 lecturas de secuenciación, el 99,95% eran lecturas limpias, mientras que sólo el 0,05% contenía contaminaciones relacionadas con adaptadores. (B) La cuantificación de la distribución de la calidad de la secuenciación a lo largo de las lecturas demostró la existencia de puntuaciones de alta calidad a lo largo de la totalidad de las lecturas, con un 94,19% de lecturas de base con una puntuación Phred superior a 30. (C) La distribución de la tasa de error a lo largo de las lecturas demostró una alta fidelidad de las lecturas con una tasa de error global del 0,03%. (D) Estrategia de estratificación de genes de miocardio utilizando 174 genes de la lista de genes del panel de cardio.
El análisis inicial informó de más de 20.000 variantes genéticas identificadas en el sujeto, en comparación con el genoma de referencia. Empleamos un paso de estratificación del riesgo de enfermedad cardiovascular para enriquecer las variantes asociadas a la enfermedad cardiovascular. Esta estratificación se realizó con 174 genes cubiertos por el panel Illumina TruSight Cardio (Pua et al., 2016). Este enfoque redujo el número de variantes a 305 (Figura 2D). De estas 305 variantes potenciales, solo una había sido anotada como de «alto riesgo». Esta variante en particular era una sustitución de un solo par de bases (G a A) dentro del intrón 3, correspondiente a c.95-108G > A de ENSFCAG00000004613. Esta variante dio lugar a la posible inclusión de un sitio aceptor de empalme de novo (Figura 3A). A continuación, utilizamos un análisis in silico para determinar la probabilidad de que esta variante intrónica actúe efectivamente como un nuevo sitio aceptor de empalme (Wang y Marín, 2006). Este método in silico identificó correctamente más del 96% de los donantes y aceptores de empalme canónicos dentro de TNNT2 (Tabla 4). Cabe destacar que el análisis in silico de la variante c.95-108G > A en el sujeto apoyó la hipótesis de que esta variante altera el empalme de TNNT2. La consecuencia de este empalme aberrante se investigó a nivel de la proteína, lo que reveló que la variante podía causar la pérdida de 223 aminoácidos terminales del carboxilo y la incorporación de sólo cinco aminoácidos nuevos, haciendo que la proteína no fuera funcional (Figura 3B).
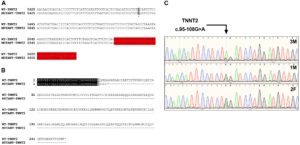
Figura 3. Análisis de la variante TNNT2 c.95-108G > A. (A) Alineación de la secuencia genética de la TNNT2 de tipo salvaje y mutante. La variante está resaltada en gris, y el exón descendente está resaltado en rojo. (B) Alineación de la secuencia proteica de la proteína TNNT2 de tipo salvaje y la proteína predicha resultante de la variante TNNT2 c.95-108G > A. (C) Cromatogramas de secuenciación Sanger que demuestran la homocigosidad para la variante TNNT2 c.95-108G > A en el sujeto y la heterocigosidad en ambos padres.
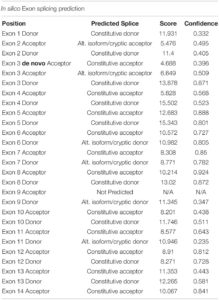
Tabla 4. Predicción in silico de los sitios de empalme de los exones del gen TNNT2 (ENSFCAG00000004613) del gato.
Se realizó la secuenciación Sanger para validar la anotación WGS de la variante c.95-108G > A en el sujeto (secuencia del cebador en la Tabla 3). Llamativamente, esta secuenciación reveló homocigosidad de la variante c.95-108G > A en el sujeto, lo que sugiere aún más su asociación con la enfermedad (Figura 3C). Tras este hallazgo, se obtuvieron muestras de sangre de ambos padres del sujeto. La ecocardiografía no sugirió ninguna patología cardíaca en ninguno de estos padres (datos no mostrados). Se extrajo el ADN y se realizó una PCR para la secuenciación Sanger de la variante TNNT2. Llamativamente, ambos padres albergaban una única copia de la variante c.95-108G > A (Figura 3C). Este hallazgo llevó a investigar el pedigrí del sujeto. Se descubrió que el sujeto había nacido de un matrimonio consanguíneo (Figura 4). En conjunto, estos resultados implican una nueva mutación intrónica que provoca el truncamiento de la proteína sarcomérica, la troponina T cardíaca, como causa de la cardiomiopatía en este gato Maine Coon.
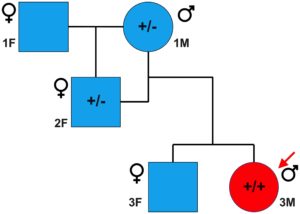
Figura 4. Pedigrí familiar del sujeto afectado. Los círculos y los cuadrados representan a los machos y las hembras, respectivamente, mientras que el azul y el rojo representan a los individuos no afectados y afectados, respectivamente. Un Maine Coon macho, heterocigoto para la variante TNNT2 c.95-108G > A, fue apareado con una Maine Coon hembra, lo que dio lugar a una cría hembra. Esta cría hembra, también heterocigótica, se cruzó con el macho original, produciendo dos crías. La cría macho fue el sujeto cardiomiopático en este estudio y era homocigoto para la variante TNNT2 c.95-108G > A. El símbolo * indica que las muestras de estos gatos no estaban disponibles en el momento del estudio.
Discusión
En este estudio, identificamos una nueva variante intrónica homocigota en TNNT2 que se asoció con un caso de cardiomiopatía felina y fallo cardíaco temprano. Se han descrito anteriormente mutaciones en MYBPC3 y MYH7. Sin embargo, hasta donde sabemos, este es el primer informe de una mutación en el filamento delgado asociada a la cardiomiopatía felina (Meurs et al., 2005; Schipper et al., 2019). Las mutaciones en TNNT2 han sido fuertemente implicadas en el desarrollo de la MCH y la miocardiopatía dilatada (Watkins et al., 1995; Pasquale et al., 2012). En humanos, estas mutaciones en TNNT2 se describen como fenotípicamente leves en comparación con las mutaciones en MYH7, pero con una mayor incidencia de muerte súbita cardíaca (Watkins et al., 1995). Es importante destacar que se han identificado mutaciones en sitios de empalme intrónicos como causantes de cardiomiopatía en humanos (Thierfelder et al., 1994). En conjunto, estos datos apoyan nuestro hallazgo de que la mutación intrónica de TNNT2 que describimos es la causa más probable de este caso de cardiomiopatía felina.
El gen TNNT2 codifica la proteína sarcomérica específica del corazón, la troponina-T cardíaca (cTnT) (Wei y Jin, 2016). La troponina cardíaca-C y -I (cTnC y cTnI, respectivamente), junto con la cTnT, constituyen el complejo de troponinas del corazón (Gomes et al., 2002). El complejo de troponina se asocia a los filamentos delgados, y se entiende que la cTnT desempeña un papel importante en el anclaje del complejo tanto a la actina como a la tropomiosina (Gomes et al., 2002). Cuando el calcio se libera del retículo sarcoplásmico, se une a la cTnC e induce un cambio estructural en ella (Vinogradova et al., 2005), lo que en última instancia conduce al movimiento de la tropomiosina, permitiendo la formación de puentes cruzados (Boussouf et al., 2007). Se ha demostrado que la cTnT regula la sensibilidad al calcio de la actomiosina ATPasa y la fuerza (Gomes et al., 2005). Así pues, la cTnT tiene un papel central en la regulación de la contracción y la relajación del corazón.
El knockout homocigótico del gen TNNT2 en ratones es embrionariamente letal (Ahmad et al., 2008; Nishii et al., 2008). Por ello, es poco probable que el sitio aceptor de empalme de novo en el TNNT2 mutante sea totalmente penetrante. Más bien, es más probable que el empalme aberrante de TNNT2 sea un evento limitado, lo que resulta en la haploinsuficiencia de la proteína cTnT. Esta variante da lugar a un truncamiento significativo de la proteína cTnT. Es bien sabido que las dos regiones carboxilo-terminales medias de la cTnT son necesarias para su interacción con la tropomiosina, la cTnI y la cTnC (Wei y Jin, 2016). Por lo tanto, la pérdida de estas regiones probablemente impediría la incorporación de la proteína truncada en el sarcómero. La reducción resultante de los niveles de cTnT daría lugar a una estequiometría anormal de las proteínas del filamento delgado en el sarcómero, lo que sería suficiente para causar una cardiomiopatía. De hecho, la haploinsuficiencia de cTnT se ha asociado previamente con la cardiomiopatía (Bollen et al., 2017). Además, la incorporación de menos del 5% de cTnT truncada en su extremo C es suficiente para causar una cardiomiopatía en ratones (Tardiff et al., 1998). Dado que los síntomas del sujeto se controlan actualmente mediante un tratamiento consistente en furosemida, espironolactona, benazepril y clopidogrel, no podemos confirmar el nivel exacto de haploinsuficiencia. Lamentablemente, no se disponía del ADN del compañero de camada del sujeto, pero prevemos que sería negativo o heterocigoto para la variante TNNT2 c.95-108G > A. Aunque no se identificaron variantes en MYH6 o MYH7, también se identificaron dos variantes de un solo nucleótido en MYBPC3 que se clasificaron como de riesgo moderado. Estas variantes eran una prolina sustituida por una leucina en la posición 922 (P922L) y una alanina por una treonina en el aminoácido 1037 (A1037T), que ocurren dentro de los dominios C7 y C8 de cMyBP-C, respectivamente. Sin embargo, una investigación más profunda de estas variantes reveló que estos aminoácidos no se conservan evolutivamente y, además, los aminoácidos sustituidos coincidían con los del ser humano o el ratón, descartando efectivamente que estas variantes de MYBPC3 fueran causantes de enfermedades.
Conclusión
Hemos identificado una nueva mutación homocigota en el gen sarcomérico TNNT2, que está asociada a una cardiomiopatía en un gato Maine Coon. La homocigosis de esta mutación es el resultado de la endogamia. Este estudio es el primero que describe una mutación en TNNT2 asociada a la cardiomiopatía felina, lo que sugiere que este gen debería incluirse en las pruebas genéticas rutinarias en los felinos. Además, los criadores deberían ser conscientes de los peligros asociados a la cría consanguínea de generaciones cercanas.
Declaración de disponibilidad de datos
Los datos de secuenciación se han depositado en la base de datos BioProject (acceso: PRJNA671288).
Declaración de Etica
El estudio del animal fue revisado y aprobado por la Ref: AM07-19-10-03-01 Institutional Animal Care and Use Committee OLAW Assurance D16-00190, AAALAC 00278 University of Cincinnati PO Box 210572, Cincinnati, OH 45267-0572. Se obtuvo el consentimiento informado por escrito del propietario para la participación de sus animales en este estudio.
Contribución del los autores
JM y SS diseñaron la investigación y redactaron el manuscrito. JM y MS realizaron la investigación. JM, MS y SS analizaron los datos. MS y RB editaron el manuscrito. RB proporcionó la supervisión del proyecto para los datos clínicos. Todos los autores leyeron y aprobaron el manuscrito final.
Financiación
La secuenciación del genoma completo del sujeto fue financiada por una donación de Kathleen y Michael Janson, propietarios del sujeto. JM recibió una beca postdoctoral de la American Heart Association (17POST33630095). SS recibió financiación de las siguientes becas: Becas de los Institutos Nacionales de Salud R01 AR078001, R01 HL130356, R56 HL139680, R01 AR067279, R01 HL105826 y R01 HL143490; y premios de transformación (19TPA34830084); y MyoKardia, AstraZeneca, Merck y Amgen.
Conflicto de Intereses
SS proporcionó estudios de consultoría e investigación en colaboración a la Fundación Leducq, Red Saree Inc, Greater Cincinnati Tamil Sangam, MyoKardia, Merck y Amgen, pero dicho trabajo no está relacionado con el contenido de este manuscrito. No se informa de ninguna otra revelación. RB forma parte de los consejos consultivos científicos de Janssen y Basking Biosciences y de los comités DSMB de Ionis Pharmaceuticals, Akcea Therapeutics y Novartis.
Los demás autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
Agradecimientos
Nos gustaría dar las gracias al personal de los hospitales veterinarios Warm Animal, de Cincinnati; Big Creek, de Cleveland; y Friendship, de Washington DC, por realizar las extracciones de sangre. También agradecemos a Linda Komar y Joseph Keyerleber por organizar los envíos de sangre de los padres del sujeto. La secuenciación Sanger fue realizada por el Centro de Secuenciación y Genotipado de ADN del Hospital Infantil de Cincinnati. Este manuscrito ha sido publicado como preimpresión en Research Square (McNamara et al., 2020).
References
Ahmad, F., Banerjee, S. K., Lage, M. L., Huang, X. N., Smith, S. H., Saba, S., et al. (2008). The role of cardiac troponin T quantity and function in cardiac development and dilated cardiomyopathy. PLoS One 3:e2642. doi: 10.1371/journal.pone.0002642
Bollen, I. A. E., Schuldt, M., Harakalova, M., Vink, A., Asselbergs, F. W., Pinto, J. R., et al. (2017). Genotype-specific pathogenic effects in human dilated cardiomyopathy. J. physiol. 595, 4677–4693. doi: 10.1113/jp274145
Boussouf, S. E., Maytum, R., Jaquet, K., and Geeves, M. A. (2007). Role of tropomyosin isoforms in the calcium sensitivity of striated muscle thin filaments. J. Muscle Res. Cell Motil. 28, 49–58. doi: 10.1007/s10974-007-9103-z
Brouwer, W. P., Van Dijk, S. J., Stienen, G. J., Van Rossum, A. C., Van Der Velden, J., and Germans, T. (2011). The development of familial hypertrophic cardiomyopathy: from mutation to bedside. Eur. J. Clin. Invest. 41, 568–578. doi: 10.1111/j.1365-2362.2010.02439.x
Depristo, M. A., Banks, E., Poplin, R., Garimella, K. V., Maguire, J. R., Hartl, C., et al. (2011). A framework for variation discovery and genotyping using next-generation DNA sequencing data. Nat. Genet. 43, 491–498.
Drourr, L., Lefbom, B. K., Rosenthal, S. L., and Tyrrell, W. D. Jr. (2005). Measurement of M-mode echocardiographic parameters in healthy adult Maine Coon cats. J. Am. Vet. Med. Assoc. 226, 734–737. doi: 10.2460/javma.2005.226.734
Ferasin, L., Sturgess, C. P., Cannon, M. J., Caney, S. M., Gruffydd-Jones, T. J., and Wotton, P. R. (2003). Feline idiopathic cardiomyopathy: a retrospective study of 106 cats (1994-2001). J. Feline Med. Surg. 5, 151–159. doi: 10.1016/s1098-612x(02)00133-x
Genova, F., Longeri, M., Lyons, L. A., Bagnato, A., Gandolfi, B., Aberdein, D., et al. (2018). First genome-wide CNV mapping in FELIS CATUS using next generation sequencing data. BMC Genom. 19:895. doi: 10.1186/s12864-018-5297-2
Gomes, A. V., Liang, J., and Potter, J. D. (2005). Mutations in human cardiac troponin I that are associated with restrictive cardiomyopathy affect basal ATPase activity and the calcium sensitivity of force development. J. Biol. Chem. 280, 30909–30915. doi: 10.1074/jbc.m500287200
Gomes, A. V., Potter, J. D., and Szczesna-Cordary, D. (2002). The role of troponins in muscle contraction. IUBMB Life 54, 323–333. doi: 10.1080/15216540216037
Ho, C. Y. (2011). New paradigms in hypertrophic cardiomyopathy: insights from genetics. Prog. Pediatr. Cardiol. 31, 93–98. doi: 10.1016/j.ppedcard.2011.02.005
Li, H., and Durbin, R. (2009). Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics 25, 1754–1760. doi: 10.1093/bioinformatics/btp324
Li, H., Handsaker, B., Wysoker, A., Fennell, T., Ruan, J., Homer, N., et al. (2009). The Sequence Alignment/Map format and SAMtools. Bioinformatics 25, 2078–2079. doi: 10.1093/bioinformatics/btp352
Longeri, M., Ferrari, P., Knafelz, P., Mezzelani, A., Marabotti, A., Milanesi, L., et al. (2013). Myosin-binding protein C DNA variants in domestic cats (A31P, A74T, R820W) and their association with hypertrophic cardiomyopathy. J. Vet. Intern. Med. 27, 275–285. doi: 10.1111/jvim.12031
McNamara, J. W., Schuckman, M., Becker, R. C., and Sadayappan, S. (2020). A novel homozygous premature stop mutation in TNNT2 associates with Feline cardiomyopathy. Res. Sq. [Preprint]. doi: 10.21203/rs.2.21647/v1
Meurs, K. M., Norgard, M. M., Ederer, M. M., Hendrix, K. P., and Kittleson, M. D. (2007). A substitution mutation in the myosin binding protein C gene in ragdoll hypertrophic cardiomyopathy. Genomics 90, 261–264. doi: 10.1016/j.ygeno.2007.04.007
Meurs, K. M., Sanchez, X., David, R. M., Bowles, N. E., Towbin, J. A., Reiser, P. J., et al. (2005). A cardiac myosin binding protein C mutation in the Maine Coon cat with familial hypertrophic cardiomyopathy. Hum. Mol. Genet. 14, 3587–3593. doi: 10.1093/hmg/ddi386
Nishii, K., Morimoto, S., Minakami, R., Miyano, Y., Hashizume, K., Ohta, M., et al. (2008). Targeted disruption of the cardiac troponin T gene causes sarcomere disassembly and defects in heartbeat within the early mouse embryo. Dev. Biol. 322, 65–73. doi: 10.1016/j.ydbio.2008.07.007
Ontiveros, E. S., Ueda, Y., Harris, S. P., and Stern, J. A. (2018). Precision medicine validation: identifying the MYBPC3 A31P variant with whole-genome sequencing in two Maine Coon cats with hypertrophic cardiomyopathy. J. Feline Med. Surg. 21:1098612×18816460.
Pasquale, F., Syrris, P., Kaski, J. P., Mogensen, J., Mckenna, W. J., and Elliott, P. (2012). Long-term outcomes in hypertrophic cardiomyopathy caused by mutations in the cardiac troponin T gene. Circ. Cardiovasc. Genet. 5, 10–17. doi: 10.1161/circgenetics.111.959973
Pua, C. J., Bhalshankar, J., Miao, K., Walsh, R., John, S., Lim, S. Q., et al. (2016). Development of a comprehensive sequencing assay for inherited cardiac condition genes. J. Cardiovasc. Transl. Res. 9, 3–11. doi: 10.1007/s12265-016-9673-5
Schipper, T., Van Poucke, M., Sonck, L., Smets, P., Ducatelle, R., Broeckx, B. J. G., et al. (2019). A feline orthologue of the human MYH7 c.5647G>A (p.(Glu1883Lys)) variant causes hypertrophic cardiomyopathy in a Domestic Shorthair cat. Eur. J. Hum. Genet. 27, 1724–1730. doi: 10.1038/s41431-019-0431-4
Tardiff, J. C., Factor, S. M., Tompkins, B. D., Hewett, T. E., Palmer, B. M., Moore, R. L., et al. (1998). A truncated cardiac troponin T molecule in transgenic mice suggests multiple cellular mechanisms for familial hypertrophic cardiomyopathy. J. Clin. Invest. 101, 2800–2811. doi: 10.1172/jci2389
Thierfelder, L., Watkins, H., Macrae, C., Lamas, R., Mckenna, W., Vosberg, H. P., et al. (1994). Alpha-tropomyosin and cardiac troponin T mutations cause familial hypertrophic cardiomyopathy: a disease of the sarcomere. Cell 77, 701–712. doi: 10.1016/0092-8674(94)90054-x
Thomas, W. P., Gaber, C. E., Jacobs, G. J., Kaplan, P. M., Lombard, C. W., Moise, N. S., et al. (1993). Recommendations for standards in transthoracic two-dimensional echocardiography in the dog and cat. Echocardiography Committee of the Specialty of Cardiology, American College of Veterinary Internal Medicine. J. Vet. Intern. Med. 7, 247–252. doi: 10.1111/j.1939-1676.1993.tb01015.x
Van Dijk, S. J., Bezold Kooiker, K., Mazzalupo, S., Yang, Y., Kostyukova, A. S., Mustacich, D. J., et al. (2016). The A31P missense mutation in cardiac myosin binding protein C alters protein structure but does not cause haploinsufficiency. Arch. Biochem. Biophys. 601, 133–140. doi: 10.1016/j.abb.2016.01.006
Vinogradova, M. V., Stone, D. B., Malanina, G. G., Karatzaferi, C., Cooke, R., Mendelson, R. A., et al. (2005). Ca(2+)-regulated structural changes in troponin. Proc. Natl. Acad. Sci. U.S.A. 102, 5038–5043. doi: 10.1073/pnas.0408882102
Viswanathan, S. K., Sanders, H. K., Mcnamara, J. W., Jagadeesan, A., Jahangir, A., Tajik, A. J., et al. (2017). Hypertrophic cardiomyopathy clinical phenotype is independent of gene mutation and mutation dosage. PLoS One 12:e0187948. doi: 10.1371/journal.pone.0187948
Wang, K., Li, M., and Hakonarson, H. (2010). ANNOVAR: functional annotation of genetic variants from high-throughput sequencing data. Nucleic Acids Res. 38:e164. doi: 10.1093/nar/gkq603
Wang, M., and Marín, A. (2006). Characterization and prediction of alternative splice sites. Gene 366, 219–227. doi: 10.1016/j.gene.2005.07.015
Watkins, H., Mckenna, W. J., Thierfelder, L., Suk, H. J., Anan, R., O’donoghue, A., et al. (1995). Mutations in the genes for cardiac troponin T and alpha-tropomyosin in hypertrophic cardiomyopathy. N. Engl. J. Med. 332, 1058–1064. doi: 10.1056/nejm199504203321603
Wei, B., and Jin, J. P. (2016). TNNT1, TNNT2, and TNNT3: Isoform genes, regulation, and structure-function relationships. Gene 582, 1–13. doi: 10.1016/j.gene.2016.01.006
Keywords: Hypertrophic Cardiomyopathy, Maine Coon, MYBPC3, TNNT2, sarcomere
Citation: McNamara JW, Schuckman M, Becker RC and Sadayappan S (2020) A Novel Homozygous Intronic Variant in TNNT2 Associates With Feline Cardiomyopathy. Front. Physiol. 11:608473. doi: 10.3389/fphys.2020.608473
Received: 20 September 2020; Accepted: 26 October 2020;
Published: 16 November 2020.
Edited by:
Xuejun Wang, University of South Dakota, United States
Reviewed by:
Michelle S. Parvatiyar, Florida State University, United States
Rongxue Wu, The University of Chicago, United States
Copyright © 2020 McNamara, Schuckman, Becker and Sadayappan. This is an open-access article distributed under the terms of the Creative Commons Attribution License (CC BY). The use, distribution or reproduction in other forums is permitted, provided the original author(s) and the copyright owner(s) are credited and that the original publication in this journal is cited, in accordance with accepted academic practice. No use, distribution or reproduction is permitted which does not comply with these terms.
*Correspondence: Sakthivel Sadayappan, sadayasl@ucmail.uc.edu
†Present address: James W. McNamara, Murdoch Children’s Research Institute, The Royal Children’s Hospital, Parkville, VIC, Australia
© 2021 Berkano Cattery – Todos los derechos reservados.
Página desarrollada con ❤ por Cattery Web.
